Stabilisation des plasmides
Les plasmides sont des éléments d'ADN le plus souvent circulaires et double brin. On les retrouve dans bon nombre de bactéries en supplément du génome bactérien de base.
Recherche sur Google Images :
Source image : fr.wikipedia.org Cette image est un résultat de recherche de Google Image. Elle est peut-être réduite par rapport à l'originale et/ou protégée par des droits d'auteur. |
Page(s) en rapport avec ce sujet :
- ) et c'est un système médical enregistré dans l'Union européenne, [1]....... germe ou un poison bien déterminé, Les porteurs de l'immunité sont .... pérennité dans une lignée de bactéries dépend par conséquent de divers moyens de stabilisation des ... La probabilité augmente avec la diminution du nombre de plasmides... (source : yonne.lautre)
Les plasmides sont des éléments d'ADN le plus souvent circulaires et double brin. On les retrouve dans bon nombre de bactéries en supplément du génome bactérien de base. Leur présence n'est a priori pas indispensable à la survie de leur cellule hôte, quoiqu'ils puissent porter des gènes codant des fonctions apportant des avantages évolutifs tels que l'exploitation d'une ressource du milieu, la synthèse d'un élément essentiel non disponible dans le milieu ou la résistance à un poison. Si tel n'est pas le cas, les plasmides représentent un poids mort de par l'utilisation des ressources cellulaires. En effet, ils sont répliqués et leurs gènes sont exprimés au détriment partiel de la réplication et de l'expression du génome propre de la cellule. En absence d'un facteur de sélection pour la présence du plasmide, une cellule fille qui ne recevrait pas de copie d'un plasmide lors de la division de sa cellule mère serait par conséquent favorisée au sein de la population.
En ce qui concerne les bactéries, on remarque que la majorité contiennent un ou plusieurs plasmides à l'état sauvage même en absence de tout facteur environnemental favorable à leur maintien. Ce maintien (ou stabilisation) d'un plasmide dans une population exploite par conséquent d'autres principes.
La réplication d'un plasmide est effectuée par la machinerie cellulaire mais l'origine de réplication et certains facteurs de réplication sont propres au plasmide. Le nombre de copies du plasmide est ainsi défini par le plasmide lui-même. Un dispositif simple de stabilisation consiste par conséquent à augmenter le nombre de copies de sorte que leur distribution aléatoire permette d'en retrouver dans chaque cellule fille après la division. En effet, la probabilité qu'une cellule se retrouve sans copie du plasmide est de 2-n où n est le nombre de copies avant la division. Ainsi, un plasmide présent en 10 copies risque d'être perdu l'ensemble des 1024 divisions. Avec 20 copies, cette probabilité tombe à moins d'une chance sur un million (probabilité toujours diminuée par l'encombrement volumique des plasmides dans la cellule). Pour rare qu'elle soit, cette perte n'en demeure pas moins un avantage évolutif sur les cellules devant toujours supporter la charge de vingt plasmides.
La stabilisation d'un plasmide à faible nombre de copies dans la population qui l'héberge nécessite d'autres mécanismes. Ces mécanismes sont de différents types et sont fréquemment cumulés sur un même plasmide pour plus d'efficacité. En voici un aperçu.
Résolution des multimères
Les différentes copies d'un plasmide possèdent la même séquence d'ADN. Elles sont par conséquent susceptibles de faire l'objet de recombinaisons homologues, formant ainsi des multimères de plusieurs plasmides. La régulation du nombre de copies se faisant selon le nombre d'origines de réplication et selon le nombre de plasmides indépendants, la multimérisation diminue le nombre d'unités totales à répartir dans les cellules filles et augmente ainsi les probabilités de produire une cellule fille sans copie plasmidique.
La solution consiste pour le plasmide à coder un dispositif de recombinase (ou à utiliser un tel dispositif présent sur le chromosome) donnant la possibilité la résolution de multimères en monomères. L'opéron parCBA du plasmide bactérien RK2 est un exemple d'un tel dispositif comprenant les gènes de la résolvase ParA, de la nucléase ParB et d'une protéine à la fonction inconnue, ParC.
Dispositifs actifs de partition
Les dispositifs de partition sont composés de séquences dont la fonction est comparable à celle des centromères eucaryotes. Certains modèles proposent que ces séquences soient liées par des protéines à des éléments uniques présents dans chaque cellule fille. Ces éléments pourraient par exemple être localisés sur le chromosome ou la membrane.
D'autres modèles proposent le pairage des plasmides par leur séquence centromérique. Ceux-ci seraient ensuite distribués dans les cellules filles soit par des filaments soit par positionnement de la paire sur le septum en formation, un plasmide de chaque côté de ce dernier.
Des exemples de dispositifs actifs de partition sont parA du plasmide R1 (à ne pas confondre avec parA de l'opéron parCBA du plasmide RK2), sop du plasmide F ou par du plasmide-prophage P1.
Dispositifs poison-antidote
Ces dispositifs sont aussi nommés module de dépendances (addiction modules), dispositifs de mort programmée ou dispositifs de post-segregational killing (ou PSK, soit tuerie «post-ségrégationnelle», non usité en français). Ils ont été identifiés sur de nombreux plasmides à faible nombre de copies dans des bactéries Gram négatives.
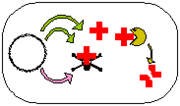
Dans un même opéron du plasmide sont codés un poison et un antidote à ce poison (figure 1. A). L'antidote est assez instable comparé au poison car rapidement dégradé par l'activité d'une protéase ATP-dépendante. Il doit par conséquent être tenu dans une concentration suffisante pour contrecarrer l'effet du poison, ce qui requiert la présence du plasmide. En effet, si, lors de la division, une cellule fille ne reçoit pas de copie du plasmide, l'antidote toujours présent dans le cytoplasme est dégradé sans qu'il ne puisse plus être synthétisé de novo. Le poison peut par conséquent agir pour tuer la cellule, d'où l'appellation de post-segregational killing (figure 1. B). Le terme «module de dépendance» fait, lui, référence au fait que la cellule est «accro» (addicted) à sa «dose» d'antidote pour survivre.
L'antidote peut être soit un ARN antisens empêchant la traduction de l'ARN messager du poison comme dans le dispositif hok/sok, soit une protéine inhibant l'action de la protéine poison par formation d'un complexe avec celle-ci. Les dispositifs protéine-protéine, outre certaines exceptions, ont des caractéristiques communes : le gène de l'antidote précède celui du poison dans l'opéron,
-
- les protéines sont de petite taille, de l'ordre de 70 à 85 acides aminés pour l'antidote et de 100 à 130 pour le poison,
- l'expression de l'antidote est plus forte que celle du poison,
- le poison est efficace à particulièrement faible dose,
- l'antidote forme un complexe fort avec le poison,
- l'antidote est dégradé par une protéase bactérienne ATP-dépendante spécifique (demi-vie de l'antidote : de 30 à 60 minutes), tandis que poison est stable,
- la de l'opéron est auto-réprimée par l'antidote ou le complexe poison-antidote,
- les prédictions de structure secondaire suggèrent la présence d'un court brin ß à l'extrémité N-terminale des antidotes, le dimère d'antidote formant ainsi un feuillet ß impliqué dans la répression par liaison spécifique à l'ADN.
Voici quelques exemples de dispositifs protéiques de poison-antidote repris dans le tableau en fin d'article :
- ccd
Ce dispositif, le mieux étudié à l'heure actuelle, est situé sur le plasmide F d'Escherichia coli. Les deux gènes de l'opéron ccd codent deux protéines : l'antidote CcdA (72 acides aminés) et le poison ccdB (101 acides aminés). Par isolement d'un mutant résistant à ccdB, il a été montré que la cible de ccdB est l'ADN gyrase, une topoisomérase de type II impliquée de la gestion du surenroulement de l'ADN. Par la suite, il a aussi été montré que ccdB empoisonne la gyrase et l'amène à provoquer des cassures double-brin dans l'ADN de manière ATP-dépendante.
L'empoisonnement de la gyrase par ccdB provoque l'induction du dispositif SOS. Ce dispositif s'enclenche lors de tout événement bloquant l'avancée de la fourche de réplication de l'ADN. Ces événements peuvent être aussi divers qu'une cassure de l'ADN, un dimère de thymine ou encore un complexe protéique avec l'ADN. L'induction de SOS bloque le processus de division cellulaire le temps que les dommages soient réparés. La cellule continuant à grandir mais ne se divisant pas, elle apparaît filamenteuse.
L'auto-répression de l'opéron ccd requiert la présence de CcdA ET de ccdB. La forme impliquée dans la liaison à l'ADN est un tétramère (CcdA) 2- (ccdB) 2, la forme libre étant un hexamère (CcdA) 2- (ccdB) 4, ce qui suggère que la baisse relative de la quantité de CcdA diminue la répression et facilite ainsi sa synthèse. CcdA41, une délétion ne comportant que les 41 acides aminés C-terminaux de CcdA, perd la capacité d'autorégulation tout en conservant ses propriétés d'anti-tuerie. Cela suggère fortement que la partie N-terminale de CcdA soit responsable de la liaison au promoteur de l'opéron ccd. Cette hypothèse est étayée par le fait qu'une mutation ponctuelle de l'arginine 4 en alanine dans CcdA abolit totalement l'activité de répression.
La protéine CcdA est dégradée par la protéase Lon malgré son affinité pour Lon particulièrement inférieure à celle pour ccdB. Les modèles proposent soit que la dégradation des formes «accidentellement» libres de CcdA soit suffisante pour provoquer le post-segregational killing, soit qu'il existe un processus actif déstabilisant le complexe CcdA-ccdB.
- pem
L'opéron pem du plasmide R100 (E. coli) code l'antidote PemI (84 acides aminés) et le poison PemK (110 acides aminés). Ce dispositif est semblable au dispositif parD du plasmide R1, l'antidote étant alors nommé Kis et le poison Kid, mais nous utiliserons la première appellation afin d'éviter toute confusion avec le dispositif parDE détaillé plus bas.
Comme ccd, la stabilisation du plasmide est due à la dégradation de l'antidote PemI par Lon et l'auto-répression complète nécessite à la fois le poison et l'antidote même si une faible répression est observée avec l'antidote seul.
La cible du poison PemK pourrait être DnaB. DnaB est une hélicase impliquée dans l'initiation de la réplication de l'ADN et une surproduction de DnaB inhibe l'action toxique de PemK). PemK agirait comme un inhibiteur de la division cellulaire même si un effet de post-segregational killing a été observé dans certaines souches.
- parDE
RK2 (appelé aussi RP4) est un grand plasmide de 60 kb (1 kb = mille paires de base d'ADN). Son spectre d'hôte est particulièrement large dans les bactéries gram–négatives avec des hôtes aussi divers qu'E. coli (entérobactérie), Agrobacterium tumefaciens (bactérie pathogène des végétaux), des rhizobia symbiotiques de légumineuses) ou Pseudomonas æruginosa (pathogène humain responsable surtout d'infections nosocomiales). Il s'y retrouve en 4 à 8 copies par chromosome selon l'espèce.
Un locus de 3, 2 kb nommé par est responsable de la stabilisation de RK2. Il contient cinq gènes organisés en deux opérons divergents. Le premier opéron, parCBA (2, 3 kb), déjà évoqué plus haut, est responsable d'un dispositif de résolution de multimères et le second, parDE (0, 7 kb), est un dispositif de post-segregational killing où ParD est l'antidote (83 acides aminés) et ParE, le poison (103 acides aminés). L'association de ces deux dispositifs permet un très faible taux de perte mais la contribution relative des deux opérons à la stabilisation est variable selon la souche d'E. coli et la température de croissance.
La cible du poison ParE n'a pas toujours été déterminée mais, comme ccdB, ParE provoque la tuerie des cellules ayant perdu le plasmide, l'inhibition de la réplication, l'induction du dispositif SOS mais aussi la naissance de bactéries filamenteuses. Sachant d'une part la diversité d'hôtes dans lesquels le dispositif parDE de RK2 est fonctionnel et , d'autre part, la difficulté à trouver des mutants résistants à ParE, on peut raisonnablement penser que ParE affecte une fonction principale et particulièrement conservée dans l'évolution, et certainement le site actif même de sa cible.
ParE est codé par un gène commençant une base en amont du codon stop de parD. Si les concentrations in vivo de ParD et ParE sont inconnues, on peut cependant estimer que l'expression de ParE est bien plus faible que celle de ParD, parE étant le second gène de l'opéron et commençant, qui plus est , par un codon TTG peu efficace au contraire des autres poisons, qui débutent par un classique codon ATG.
L'auto-répression est accomplie par ParD ou par le complexe ParD-ParE, sans qu'on ait pu déceler une quelconque action de ParE dans cette répression. Si ParD se trouve sous forme de dimère en solution, ce sont cependant 4 molécules de ParD qui se fixent au promoteur PparDE.
La structure 3D de ParD est inconnue mais les prédictions de structure secondaire convergent pour dire qu'elle contient un brin ß N-terminal suivi de 3 ou 4 hélices a connectées par de courtes boucles. Ces résultats concordent avec les études de spectroscopie par dichroïsme circulaire et par résonance magnétique nucléaire. ParD rejoindrait ainsi la famille des protéines de liaison à l'ADN à feuillets ß.
De la même manière que pour CcdA et que pour PemI, la protéase d'E. coli responsable de l'instabilité relative de ParD est Lon).
- mazEF et autres dispositifs chromosomiques
Le module mazEF (ou chpA) est localisé dans l'opéron rel en aval du gène codant la protéine RelA. Contrairement aux qui ont précédé, il ne se situe pas sur un plasmide mais dans le chromosome même dE. coli. Ce n'est par conséquent pas un dispositif de post-segregational killing car, contrairement aux plasmides, le chromosome ne se «perd» pas… Sa séquence est partiellement homologue à celle de pem ainsi qu'à un autre dispositif chromosomique dE. coli, chpB. L'expression de mazEF (MazE, 82 acides aminés, étant l'antidote et MazF, 111 acides aminés, le poison) est inhibée par la guanosine-3', 5'-bispyrophosphate (ppGpp) synthétisée par RelA lors de carences extrêmes en acides aminés. Dans ce cas, MazE étant dégradé par la protéase ClpPA, MazF est libre d'empoisonner sa cible (inconnue), menant ainsi à la mort cellulaire. On pense que cela permettrait la survie de la population bactérienne par sacrifice de bon nombre des cellules carencées.
Il a d'autre part été montré que ce dispositif de mort programmée pouvait être induit par des antibiotiques inhibiteurs de la ou de la traduction tels la rifampicine, le chloramphénicol ou la spectinomycine. Il est aussi activé par le dispositif phd/doc dont nous parlerons peu après.
Un autre dispositif chromosomique, relBE, a été découvert dans le génome d'E. coli K12 et est homologue à plusieurs dispositifs plasmidiques ou chromosomiques dE. coli ou d'autres espèces bactériennes gram–négatives, ainsi qu'à des dispositifs chromosomiques chez des gram-positives et même des archées. À ce jour, aucun homologue n'a été trouvé chez les eucaryotes mais il a été montré que RelE dE. coli est aussi actif dans Saccharomyces cerevisiæ et que RelB en contrecarre l'action, du moins partiellement, le taux d'expression de RelB étant assez bas dans la procédure utilisée.
On retrouve aussi des homologues chromosomiques de ccd dans la souche pathogène O157 :H7 d'E. coli, de hok/sok dans E. coli ou, toujours, de parDE dans Mycobacterium tuberculosis, Vibrio choleræ ou Yersinia enterocolitica.
- phd/doc
Dans sa phase lysogène, le bactériophage P1 d'E. coli est présent sous forme d'un plasmide à faible nombre de copies. Il contient le module de PSK phd/doc où Phd (73 acides aminés) est l'antidote instable et Doc (126 acides aminés), le poison stable. Phd est dégradé par la protéase ClpPX. Phd est capable d'autoréguler l'opéron mais est plus efficace en présence de Doc.
La cible de Doc n'est pas toujours connue mais elle serait impliquée dans une étape principale pour la synthèse des protéines. Il a été établi que la présence de mazEF était requise pour la fonction post-segregational killing de phd/doc, suggérant que Doc provoque l'inhibition de la traduction de l'antidote MazE, menant ainsi à la mort. Ces deux mécanismes seraient par conséquent couplés en cascade.
- restriction-modification
Un autre type de dispositif proche des poison-antidote est le dispositif restriction-modification (RM). D'ordinaire, un des dispositifs bactériens pour se protéger des ADN étrangers (un phage, par exemple) est la présence d'enzymes de restriction qui clivent la molécule d'ADN sur ou à proximité d'un site spécifique. La protection du génome de la bactérie est assurée par des enzymes de modification méthylant la molécule d'ADN, empêchant ainsi l'action des enzymes de restriction.
Ce dispositif fonctionne aussi pour la stabilisation de plasmides. La différence principale avec les dispositifs «classiques» poison-antidote tient dans ce que les deux enzymes ne forment pas un complexe inactivant l'enzyme de restriction. Qui plus est , l'effet de post-segregational killing ne serait pas dû à une dégradation plus rapide de la méthylase donnant la possibilité ainsi la libération du poison. Il a ainsi été proposé un modèle où la dilution progressive des deux enzymes lors des divisions successives mènerait à un point où la méthylation n'est plus assez performante, laissant le peu d'enzymes de restriction restantes causer des dommages irréparables à l'ADN.
Tableau : Propriétés de dispositifs poison-antidote
Dispositif Localisation Antidote, nombre d'acides aminés Poison, nombre d'acides aminés Cible du poison Protéase dégradant l'antidote ccd plasmide F CcdA, 72 CcdB, 101 gyrase Lon pem plasmides R1 et R100 PemI (ou Kis), 84 PemK (ou Kid), 110 DnaB ? * Lon parDE plasmide RK2 (ou RP4) ParD, 83 ParE, 103 non déterminée Lon mazEF (ou chpA) chromosome d'E. coli MazE (ou ChpAI), 82 MazF (ou ChpAK), 111 non déterminée ClpAP phd/doc Plasmide P1 Phd, 73 Doc, 126 non déterminée ClpXP
* : aucun mutant résistant à PemK n'a été obtenu mais la surproduction de DnaB inhibe l'action de PemK, ce qui suggère que DnaB soit la cible de PemK.
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 20/03/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité